Vì vậy, tôi đã tạo ra một giải pháp khác bằng cách sử dụng các phần của cả giải pháp yuk và gnovice. Khi tôi chơi xung quanh với các giải pháp tôi nhận ra tôi thực sự muốn có thể sử dụng đầu ra là "subplots" và có thể thay đổi màu sắc của các chữ cái tùy ý.
Vì yuk sử dụng các đối tượng trục được đặt chương trình với chữ nhúng nó sẽ rất khó chịu (mặc dù không thể) để sửa đổi mã của mình thành ô đối tượng trục tùy ý. Kể từ khi giải pháp của gnovice đọc các chữ cái từ một tập tin được tạo trước nó sẽ có khó khăn để sửa đổi mã để chạy chống lại các chương trình màu tùy ý hoặc lựa chọn thư. Vì vậy, giải pháp của tôi sử dụng mã "tạo thư" từ giải pháp của yuk và phương pháp "siêu hình ảnh" từ giải pháp của gnovice.
Ngoài ra còn có một số lượng đáng kể phân tích đối số và kiểm tra. Dưới đây là giải pháp kết hợp của tôi ... Tôi chỉ bao gồm nó để hoàn thành, tôi rõ ràng không thể giành chiến thắng tiền thưởng của riêng tôi. Tôi sẽ để cho cộng đồng quyết định giải thưởng và trao tiền thưởng cho bất cứ ai có xếp hạng cao nhất vào cuối thời hạn ... trong trường hợp hòa tôi sẽ đưa nó cho người có người đại diện thấp nhất (họ có lẽ "cần" nó nhiều hơn).
function [npos, handle] = SeqLogoFig(SEQ, varargin)
% SeqLogoFig
% A function which wraps around the bioinformatics SeqLogo command
% and creates a figure which is actually a MATLAB figure. All
% agruements for SEQLOGO are passed along to the seqlogo calculation.
% It also supports extra arguements for plotting.
%
% [npos, handle] = SeqLogoFig(SEQ);
%
% SEQ A multialigned set of sequences that is acceptable
% to SEQLOGO.
% npos The positions that were actually plotted
% handle An axis handle to the object that was plotted.
%
% Extra Arguements:
%
% 'CUTOFF' A bit-cutoff to use for deciding which columns to
% plot. Any columns that have a MAX value which is
% greater than CUTOFF will be provided. Defaults to
% 1.25 for NT and 2.25 for AA.
%
% 'TOP-N' Plots only the top N columns as ranked by thier MAX
% bit conservation.
%
% 'AXES_HANDLE' An axis handle to plot the seqlogo into.
%
% 'INDS' A set of indices to to plot. This overrides any
% CUTOFF or TOP-N that were provided
%
%
%
%
%% Parse the input arguements
ALPHA = 'nt';
MAX_BITS = 2.5;
RES = [200 80];
CUTOFF = [];
TOPN = [];
rm_inds = [];
colors = [];
handle = [];
npos = [];
for i = 1:2:length(varargin)
if strcmpi(varargin{i}, 'alphabet')
ALPHA = varargin{i+1};
elseif strcmpi(varargin{i}, 'cutoff')
CUTOFF = varargin{i+1};
%we need to remove these so seqlogo doesn't get confused
rm_inds = [rm_inds i, i+1]; %#ok<*AGROW>
elseif strcmpi(varargin{i}, 'colors')
colors = varargin{i+1};
rm_inds = [rm_inds i, i+1];
elseif strcmpi(varargin{i}, 'axes_handle')
handle = varargin{i+1};
rm_inds = [rm_inds i, i+1];
elseif strcmpi(varargin{i}, 'top-n')
TOPN = varargin{i+1};
rm_inds = [rm_inds i, i+1];
elseif strcmpi(varargin{i}, 'inds')
npos = varargin{i+1};
rm_inds = [rm_inds i, i+1];
end
end
if ~isempty(rm_inds)
varargin(rm_inds) = [];
end
if isempty(colors)
colors = GetColors(ALPHA);
end
if strcmpi(ALPHA, 'nt')
MAX_BITS = 2.5;
elseif strcmpi(ALPHA, 'aa')
MAX_BITS = 4.5;
end
if isempty(CUTOFF)
CUTOFF = 0.5*MAX_BITS;
end
%% Calculate the actual seqlogo.
wm = seqlogo(SEQ, varargin{:}, 'displaylogo', false);
%% Generate the letters
letters = wm{1};
letter_wins = cell(size(letters));
[~, loc] = ismember(letters, colors(:,1));
loc(loc == 0) = size(colors,1);
clr = cell2mat(colors(loc, 2)); % corresponding colors
for t = 1:numel(letters)
hf = figure('position',[200 200 100 110],'color','w');
ha = axes('parent',hf, 'visible','off','position',[0 0 1 1]);
ht = text(50,55,letters(t),'color',clr(t,:),'units','pixels',...
'fontsize',100,'fontweight','norm',...
'vertical','mid','horizontal','center');
F = getframe(hf); % rasterize the letter
img = F.cdata;
m = any(img < 255,3); % convert to binary image
m(any(m,2),any(m,1))=1; % mask to cut white borders
letter_wins{t} = reshape(img(repmat(m,[1 1 3])),[sum(any(m,2)) sum(any(m,1)) 3]);
close(hf);
end
%% Use the letters to generate a figure
%create a "image" that will hold the final data
wmat = wm{2};
if isempty(npos)
if isempty(TOPN)
npos = find(any(wmat>CUTOFF,1));
else
[~, i] = sort(max(wmat,[],1), 'descend');
npos = sort(i(1:TOPN));
end
end
fig_data = 255*ones(RES(1), RES(2)*(length(npos)+1)+length(npos)*2,3);
bitscores = linspace(0, MAX_BITS, size(fig_data,1));
tick_pos = zeros(length(npos),1);
% place images of letters
for i=1:length(npos)
[wms idx] = sort(wmat(:,npos(i)), 'descend'); % largest on the top
bits = [flipud(cumsum(flipud(wms))); 0];
let_data = letter_wins(idx(wms>0));
for s=1:length(let_data)
start_pos = find(bitscores>=bits(s),1);
end_pos = find(bitscores<=bits(s+1),1, 'last');
if isempty(start_pos) || isempty(end_pos) || end_pos > start_pos
continue
end
img_win = imresize(let_data{s}, [start_pos-end_pos, RES(2)]);
fig_data(start_pos-1:-1:end_pos, (i*RES(2)-RES(2)*.5:i*RES(2)+RES(2)*.5-1)+2*i,:) = img_win;
end
tick_pos(i) = i*RES(2)+2*i;
end
if ~isempty(handle)
image(handle,[0 size(fig_data,2)], [0 MAX_BITS],fig_data./255)
else
handle = image([0 size(fig_data,2)], [0 MAX_BITS],fig_data./255);
end
set(gca, 'ydir', 'normal', 'xtick', tick_pos, ...
'userdata', tick_pos, 'xticklabel', npos);
xlabel('position')
ylabel('bits')
function colors = GetColors(alpha)
% get the standard colors for the sequence logo
if strcmpi(alpha, 'nt')
colors = cell(6,2);
colors(1,:) = {'A', [0 1 0]};
colors(2,:) = {'C', [0 0 1]};
colors(3,:) = {'G', [1 1 0]};
colors(4,:) = {'T', [1 0 0]};
colors(5,:) = {'U', [1 0 0]};
colors(6,:) = {'', [1 0 1]};
elseif strcmpi(alpha, 'aa')
colors = cell(21,2);
colors(1,:) = {'G', [0 1 0]};
colors(2,:) = {'S', [0 1 0]};
colors(3,:) = {'T', [0 1 0]};
colors(4,:) = {'Y', [0 1 0]};
colors(5,:) = {'C', [0 1 0]};
colors(6,:) = {'Q', [0 1 0]};
colors(7,:) = {'N', [0 1 0]};
colors(8,:) = {'A', [1 165/255 0]};
colors(9,:) = {'V', [1 165/255 0]};
colors(10,:) = {'L', [1 165/255 0]};
colors(11,:) = {'I', [1 165/255 0]};
colors(12,:) = {'P', [1 165/255 0]};
colors(13,:) = {'W', [1 165/255 0]};
colors(14,:) = {'F', [1 165/255 0]};
colors(15,:) = {'M', [1 165/255 0]};
colors(16,:) = {'D', [1 0 0]};
colors(17,:) = {'E', [1 0 0]};
colors(18,:) = {'K', [0 0 1]};
colors(19,:) = {'R', [0 0 1]};
colors(20,:) = {'H', [0 0 1]};
colors(21,:) = {'', [210/255 180/255 140/255]};
else
error('SeqLogoFigure:BADALPHA', ...
'An unknown alphabet was provided: %s', alpha)
end
Tôi đã gửi tệp này đến Mathworks FileExchange ... khi được phê duyệt, tôi sẽ đăng liên kết.
Nỗi phiền toái dai dẳng duy nhất mà tôi có là vì nó tạo ra hình ảnh chữ, nó hiển thị các cửa sổ hình nhỏ với tốc độ nhanh. Nếu có ai biết một mẹo có thể tránh được thì tôi rất muốn nghe nó.
EDIT: Mathworks đã phê duyệt tệp đã gửi của tôi ...bạn có thể tải xuống tại FileExchange tại đây: http://www.mathworks.com/matlabcentral/fileexchange/27124
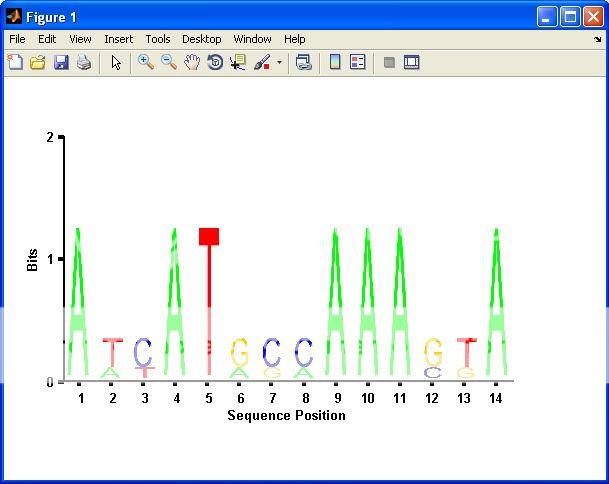
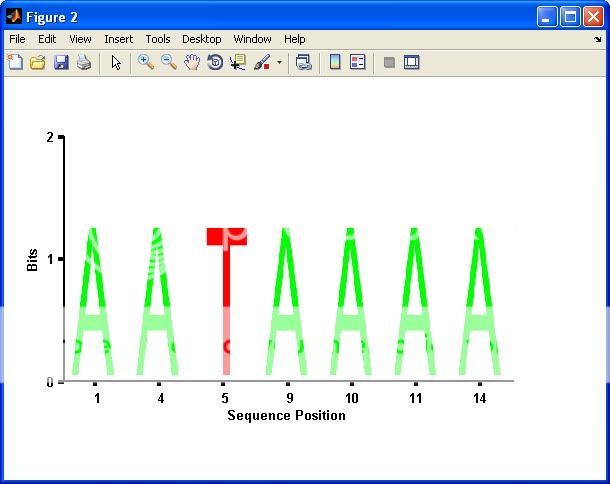
@gnoice và @yuk Tôi đã lấy một phần câu trả lời của gnovice và một câu trả lời của gnovice. Tôi sử dụng phần "tạo thư" của yuk cho các phân đoạn hình ảnh ... vì đôi khi tôi làm AA và đôi khi NT quan trọng để có thể thực hiện điều này một cách nhanh chóng. Tuy nhiên, thay vì viết chúng vào các tập tin tôi chỉ lưu các hình ảnh trong một ô. Sau đó, tôi sử dụng phương pháp của gnovice viết các tập tin hình ảnh vào một ma trận hình ảnh khổng lồ ... mặc dù tôi sử dụng IMRESIZE để thay đổi kích thước hình ảnh. Câu trả lời cuối cùng của tôi là gần 50% mã từ mỗi cộng với một số phân tích cú pháp arg và kiểm tra. Tôi không chắc chắn làm thế nào để giải thưởng "danh tiếng", bất kỳ suy nghĩ bạn hai? – JudoWill
Nếu bạn đang gặp khó khăn trong việc lựa chọn một, tôi cho rằng bạn chỉ có thể cho phép cộng đồng bỏ phiếu cho các câu trả lời trong một vài ngày, sau đó chọn phiếu bầu được bầu chọn cao nhất. Bạn * có thể * đăng giải pháp kết hợp của bạn như một câu trả lời vì lợi ích của sự hoàn chỉnh, nhưng bạn sẽ không thể tự thưởng cho mình * tiền thưởng cho nó (http://meta.stackexchange.com/questions/18841/lost- danh tiếng-sau-trả lời-của tôi-riêng-câu hỏi-với-bounty). – gnovice
@gnovice: Đó là kế hoạch của tôi (cho cả giải thưởng và tính hoàn chỉnh) ... Tôi cũng đang tạo ra một giải pháp "tổng quát" hơn và đưa nó lên MathWorks FileExchange ... Tôi sẽ đảm bảo rằng tôi tham khảo bài đăng này:) – JudoWill